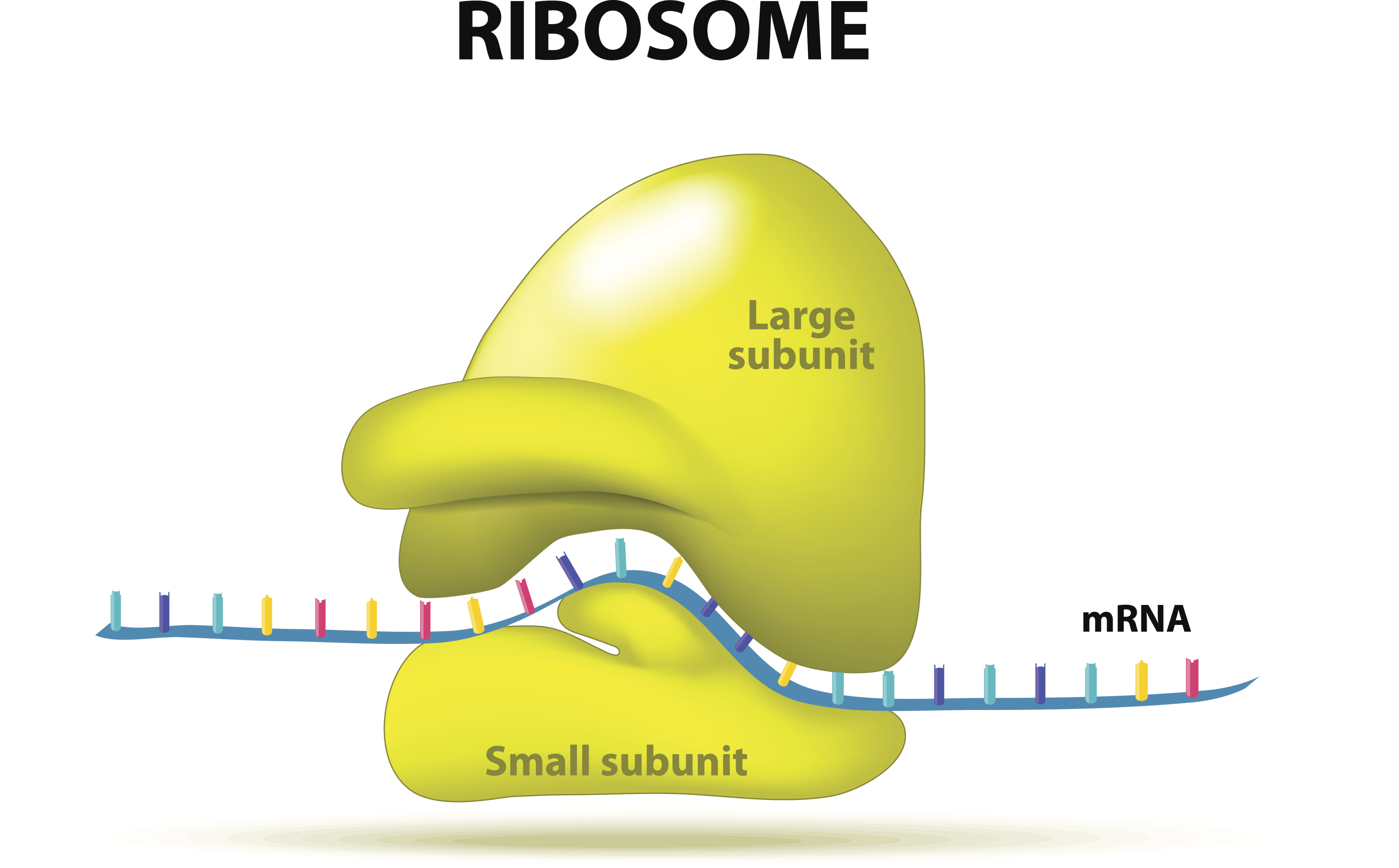
 Schematische Darstellung der Translation der mRNA am Ribosom. © ttsz/iStock/Thinkstock
Schematische Darstellung der Translation der mRNA am Ribosom. © ttsz/iStock/ThinkstockKlassische Methoden zur Erfassung von Genregulationen dokumentieren nur das Anschalten und Ablesen von Genen. Wird ein Gen abgelesen, wird sein in der Sprache der DNA enthaltener Bauplan für Proteine im Zellkern in RNA umgeschrieben (transkribiert). „Auf dieser Ebene erkennt man viele aber bei weitem nicht alle individuellen Unterschiede in der Genregulation“, erläutert Prof. Norbert Hübner, Senior Autor der Publikation und Leiter der Forschungsgruppe Medizinische Genomforschung und Genetik von Herz-Kreislauferkrankungen im MDC.
Zusammen mit Sebastian Schafer (MDC, NHRIS) und Eleonora Adami (MDC) sowie Forschern aus mehreren Forschungseinrichtungen in Berlin, den Niederlanden, England und Tschechien, untersuchten sie deshalb die Genregulation auf der nächsten Ebene, der Translation. Sie findet außerhalb des Zellkerns, im Zellplasma statt. Bei der Translation wird die RNA-Sequenz in Aminosäuresequenzen übertragen (translatiert) und in den Proteinfabriken der Zelle, den Ribosomen, zu Proteinen zusammengebaut.
Translation reguliert Herz- und Lebergene
Zunächst durchforsteten die Wissenschaftler das komplette Genom zweier Rattenstämme – ein Stamm hatte Bluthochdruck, der andere nicht. Dort schauten sich speziell die Gene von Herz- und Lebergewebe an. Dann setzen sie eine neue Technik ein, die als Ribosomal-profiling bezeichnet wird, kurz Ribo-seq. Sie ermöglicht es festzustellen, welcher Anteil an RNA tatsächlich in Protein umgesetzt (translatiert) wird.
Das Ergebnis: Bei der Translation konnten die Forscher fast doppelt so viele unterschiedlich angeschaltete Herz- und Lebergene erfassen, als bei der Transkription. Anschließend verglichen sie diese Daten mit den entsprechenden Genen des Menschen in genomweiten Assoziierungsstudien. Hier zeichnet sich ab, dass offenbar viele Herz- und Lebergene des Menschen hauptsächlich während der Translation reguliert werden. Die Forscher sind davon überzeugt, dass das Erfassen individueller Unterschiede bei der Translation neue Einblicke in die Genregulation von Erkrankungen ermöglicht.
Weitere Informationen:
Originalstudie im Fachjournal Nature Communications (doi: 10.1038/ncomms8200)