Die Forscher haben herausgefunden, dass im Lebenszyklus dieser Viren während des Zusammenbaus (Assemblierung) der infektiösen Viruspartikel neben den Strukturproteinen auch die Nichtstrukturproteine (NS) sowie verschiedene zelluläre Komponenten involviert sind. Außerdem bilden die Nichtstrukturproteine die viralen Replikationskomplexe aus.
Diese Prozesse müssen exakt reguliert werden, um die zeitliche Abfolge der einzelnen Stadien des viralen Lebenszyklus zu gewährleisten. Diese Regulation beruht auf der Assemblierung verschiedener viraler Subkomplexe mit unterschiedlichen Funktionen entweder in der Genomreplikation oder der sich daran anschließenden Bildung der Viruspartikel.
Erstmals Struktur ermittelt
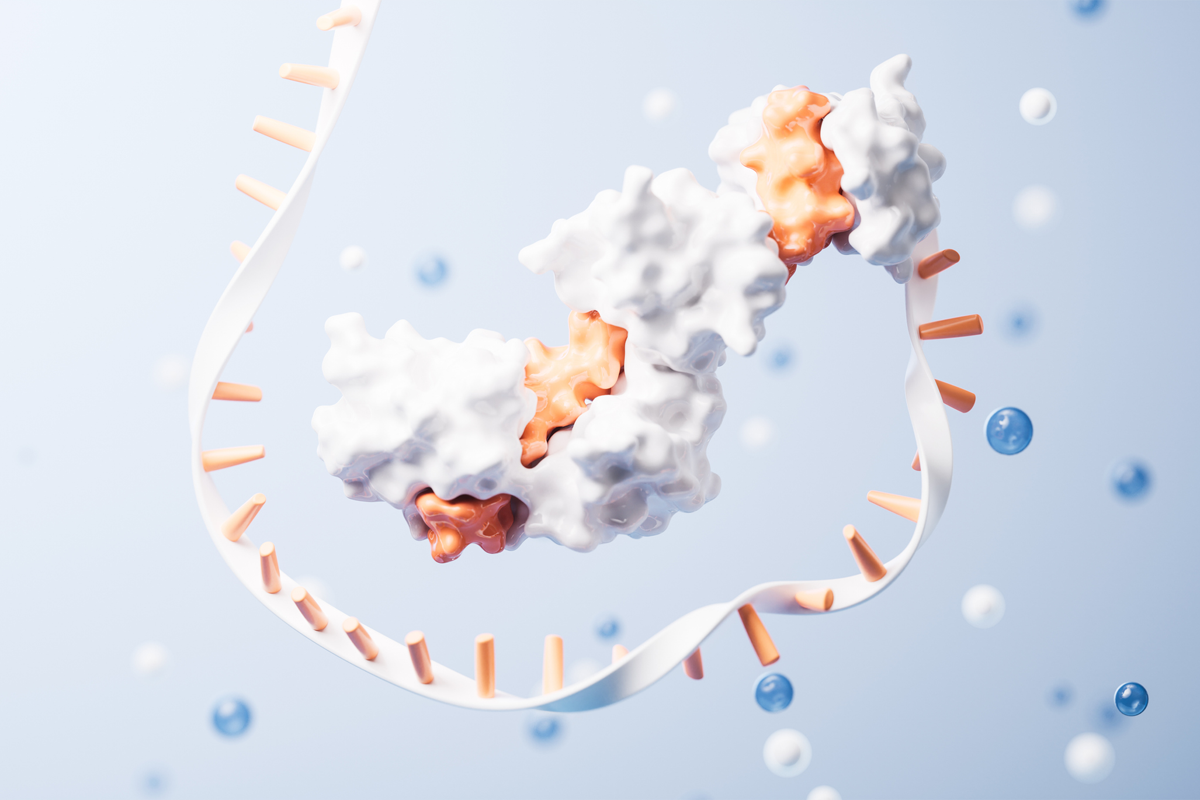
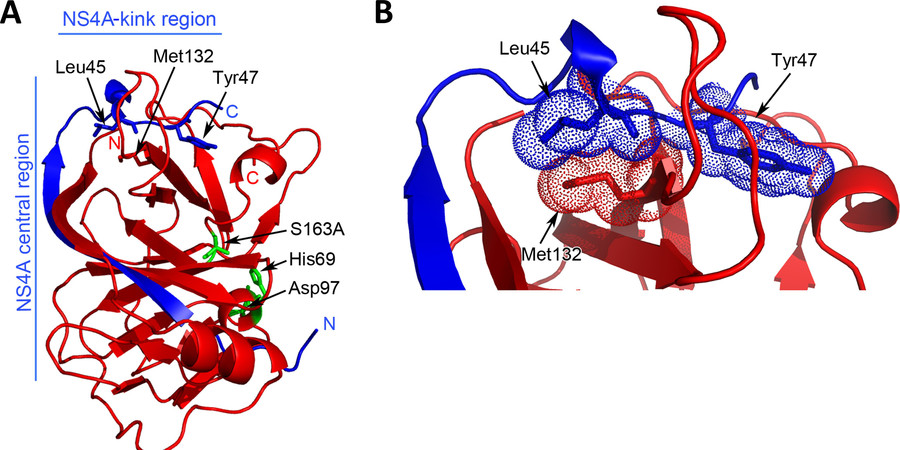 (A) 3D-Struktur der CSFV NS3 Proteasedomäne (rot) im Komplex mit ihrem Kofaktor NS4A (blau). (B) Vergrößerte Darstellung der hydrophoben Interaktionsfläche der NS3/NS4A-kink Region; als Sphären hervorgehoben sind die interagierenden Aminosäuren NS3/Met132 sowie NS4A/Leu45 and 4A/Tyr47. © Universität zu Lübeck
(A) 3D-Struktur der CSFV NS3 Proteasedomäne (rot) im Komplex mit ihrem Kofaktor NS4A (blau). (B) Vergrößerte Darstellung der hydrophoben Interaktionsfläche der NS3/NS4A-kink Region; als Sphären hervorgehoben sind die interagierenden Aminosäuren NS3/Met132 sowie NS4A/Leu45 and 4A/Tyr47. © Universität zu LübeckDie detaillierte Charakterisierung dieser Regulation wurde durch eine Kombination aus strukturellen und molekularbiologischen Untersuchungen möglich. Bei dieser Arbeit kooperierten Forscher aus Lübeck um Prof. Norbert Tautz (Institut für Virologie und Zellbiologie der Universität zu Lübeck) mit Forschern aus dem Institut Pasteur (Paris).
In der Studie konnte zum ersten Mal eine Struktur des funktionellen NS3/4A-Komplexes ermittelt werden, welche detailliert die Oberflächeninteraktion zwischen diesen beiden Proteinen zeigt. Mutationen an diesen interagierenden Oberflächen belegten eine regulatorische Rolle dieser Protein-Protein Kontakte.
Während der Kontakt zwischen NS3 und der NS4A-kink Region essentiell für die virale RNA-Replikation ist, stellt eine offene räumliche Anordnung (Konformation) des Komplexes eine Voraussetzung für die Bildung einzelner infektiöser Viruspartikel außerhalb einer Zelle (Virionen) dar.
Effektive Nutzung von Proteinrepertoire
„Wir zeigen in dieser Arbeit, dass das multifunktionelle NS3-Protein alternative Protein-Kontakte mit NS4A innerhalb verschiedener Subkomplexe eingeht und dadurch ein Umschalten von der Genomreplikation zur Bildung infektiöser Viruspartikel ausgelöst werden kann“, erläutert Prof. Tautz.
„Unsere Ergebnisse sind ein exzellentes Beispiel dafür, wie Viren ihr begrenztes Proteinrepertoire effektiv nutzen, um eine geordnete Abfolge der verschiedenen Stadien des viralen Lebenszyklus zu gewährleisten.“
Quelle: Universität zu Lübeck
Publikation: Norbert Tautz et al.; A positive-strand RNA virus uses alternative protein-protein interactions within a viral protease/cofactor complex to switch between RNA replication and virion morphogenesis; PLoS Pathog., 2017; doi: 10.1371/journal.ppat.1006134