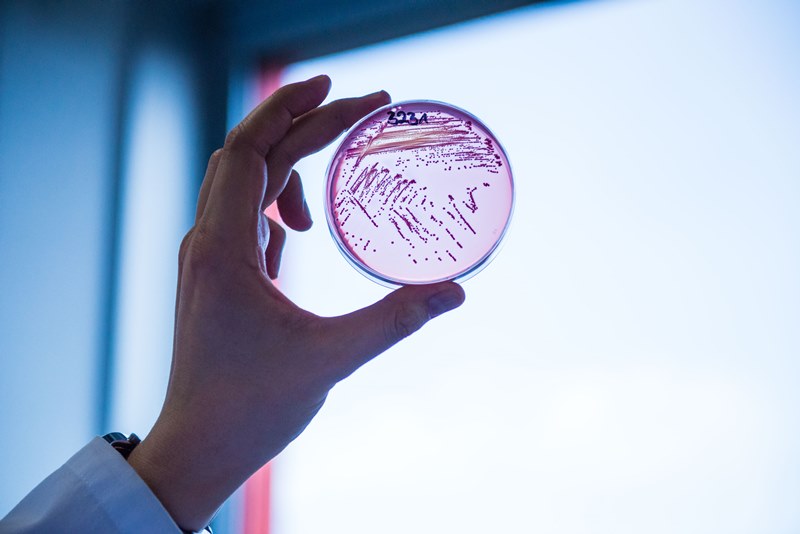
Escherichia coli, kurz E. coli genannt, gehört zu den Gram-negativen Enterobakterien, die vor allem im menschlichen Darm zu Hause sind. Einige Stämme können Infektionen auslösen, wenn sie in den übrigen Körper gelangen. Insbesondere bei geschwächten Patienten kann es zu Blutstrominfektionen, Wund- oder Harnwegsinfekten kommen.
Ihre Behandlung wird zunehmend schwerer, denn im Kampf gegen Antibiotika haben E. coli-Bakterien und andere Enterobakterien einen Abwehrmechanismus entwickelt: Sie bilden Enzyme aus, die Antibiotika unwirksam machen können: die sog. Beta-Laktamasen mit erweitertem Spektrum (ESBL). Durch ihren Mechanismus werden die bakteriellen Erreger multiresistent und sind in Kliniken besonders gefürchtet.
„Wir müssen besonders eine Untergruppe eines multiresistenten E. coli-Bakteriums im Blick behalten, die wir in unserer aktuellen Studie gefunden haben“, erklärt Prof. Trinad Chakraborty, Direktor des Instituts für Medizinische Mikrobiologie an der Justus-Liebig-Universität in Gießen und Koordinator am DZIF-Standort Gießen-Marburg-Langen. Diese Untergruppe breitet sich momentan weltweit aus und wurde nun auch in Deutschland gefunden.
Genomsequenzierung zur Überwachung von Antibiotikaresistenzen
Die Gießener Wissenschaftler untersuchten in ihrer Studie insgesamt fast 1000 Isolate von ESBL-produzierenden Bakterien aus Mensch, Tier, Umwelt und Lebensmitteln. Ihr Vorgehen ist dabei ganz im Sinn des One Health-Ansatzes, der nicht nur den Menschen, sondern auch seine Umwelt mit in die Untersuchungen einbezieht.
Dabei identifizierten sie gezielt die Gene für die Beta-Laktamasen und suchten nach einer Untergruppe, die in anderen Ländern bereits auf dem Vormarsch ist. Es handelt sich hierbei um einen multiresistenten E. coli-Stamm vom Sequenztyp 131 (ST131), der weltweit für Millionen von Infektionen verantwortlich ist, vor allem Blutstrominfektionen und Blasenentzündungen, und der ein relativ seltenes ESBL-Gen trägt, nämlich blaCTX-M-27.
Die Suche war erfolgreich: Die Forscher fanden E. coli ST131 CTX-M27 ausschließlich in menschlichen Isolaten und konnten nachweisen, dass seine Häufigkeit von Prozent im Jahr 2009 auf 45 Prozent 2016 gestiegen ist. „Damit macht dieser E. coli-Stamm mit seinem spezifischen ESBL-Gen einem E. coli ST131-Stamm Konkurrenz, der bisher in Deutschland am häufigsten nachgewiesen wurde und ein anderes ESBL-Gen trägt“, erklärt Dr. Can Imirzalioglu, Wissenschaftler der Uni Gießen.
Zum Projekt
Hier sind weitere Studien notwendig, um die Ursachen und die klinische Bedeutung dieses Shifts zu untersuchen. Dennoch zeigen die Ergebnisse, wie wichtig der Einsatz von modernen Verfahren wie der Genomsequenzierung ist, um solche Entwicklungen zu beobachten und notfalls schnell reagieren zu können.
Das Projekt wurde vom DZIF und vom Forschungsverbund RESET unterstützt, der sich der Untersuchung von Resistenzen gegen Antibiotika in Enterobakterien verschrieben hat. Seit 2013 arbeiten Wissenschaftler des DZIF-Schwerpunkts „Krankenhauskeime und Antibiotika-resistente Bakterien“ mit dem Forschungsverbund RESET in Gießen eng zusammen bei der Sammlung multiresistenter Infektionserreger von Tier und Mensch.
Das Erbgut der Isolate dieser umfangreichen Sammlung wurde in der Bioinformatischen Abteilung des DZIF sequenziert und die bakteriellen Genome in einer Datenbank hinterlegt. Diese Genom-Datenbank wird immer wieder erfolgreich genutzt, um das Vorkommen von Resistenzen näher zu untersuchen.
Quelle: Deutsches Zentrum für Infektionsforschung (DZIF)
Publikation: Trinad Chakraborty et al.; blaCTX-M-27–Encoding Escherichia coli Sequence Type 131 Lineage C1-M27 Clone in Clinical Isolates; Germany Emerging Infectious Diseases, 2017