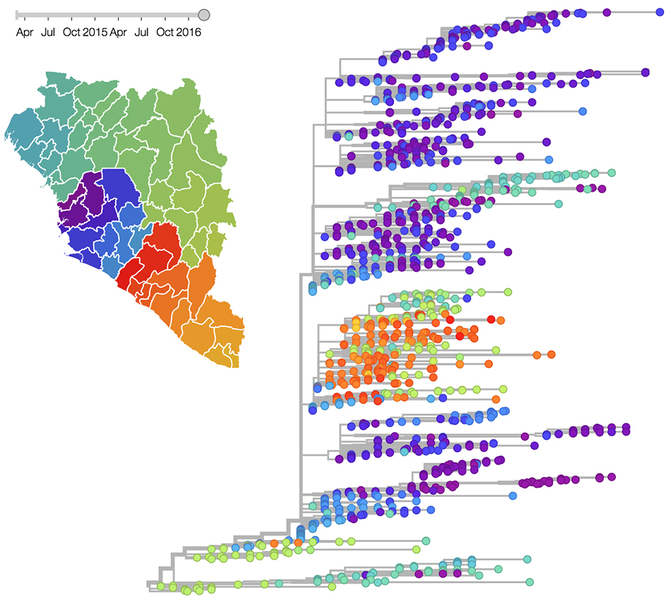
 Rekonstruierter Stammbaum von Ebola-Viren mit dazugehöriger geographischer Ausbreitung der Infektion. © R.Neher / T.Bedford
Rekonstruierter Stammbaum von Ebola-Viren mit dazugehöriger geographischer Ausbreitung der Infektion. © R.Neher / T.BedfordVirusinfektionen sind ein weltweites Gesundheitsproblem. Influenzaviren verursachen geschätzt zwischen 250 000 und 500 000 Todesfälle pro Jahr und das derzeit kursierende Zikavirus ist verantwortlich für zehntausende Fehlbildungen bei Ungeborenen. Gesundheitsbehörden können nur effektiv intervenieren, wenn ausreichende Informationen zur Epidemiologie der Viren bekannt sind.
Die Analyse von Genomsequenzen kann die Evolution und Epidemiologie solcher Viren aufdecken. Dazu müssen aber verschiedene Gruppen, Wissenschaftler oder Krankenhäuser zeitnah ihre Sequenzdaten zur Verfügung stellen, denn nur aus vielen Daten kann ein aktuelles Gesamtbild berechnet werden.
Dr. Neher und Dr. Bedford wollen daher eine Online-Plattform schaffen, die einen schnellen Austausch und die Analyse der Daten ermöglicht. Sie haben eine Software entwickelt, die den Verwandtschaftsgrad der Viren anhand der spontanen Mutationen, die bei Viren auftreten, rekonstruiert. Daraus lässt sich ein Stammbaum erstellen, der den aktuellen Verlauf einer Infektion abbildet.
„Die Software kombiniert Daten verschiedener Orte und Forschungsgruppen. Die Genomsequenz einer neuen Probe kann innerhalb von 24 Stunden in unsere Auswertung integriert werden“, sagt Neher. Für das Influenzavirus haben die beiden Wissenschaftler schon eine solche Software entwickelt: www.nextflu.org. Jetzt wollen sie diese auch für andere Viren erweitern.
Die neue Plattform wird über www.nextstrain.org öffentlich verfügbar sein. Der Quellcode unterliegt einer freien Dokumentationslizenz, sodass die Software auch von anderen Wissenschaftlern genutzt und in Zukunft für weitere Krankheiten angepasst werden kann.
Mutationen identifizieren
Über die Plattform können Gesundheitsbehörden dann wichtige und aktuelle Informationen zur Ausbreitung einer Virusinfektion abrufen. Mithilfe der Software können die Verantwortlichen nachvollziehen, in welchen geographischen Regionen sich die Viren gehäuft vermehren und welche Übertragungsketten es gibt. So können sie gezielte Maßnahmen zur Eindämmung von Infektionen treffen, etwa durch ein Vorgehen gegen Mücken, die die Viren übertragen.
Außerdem kann die Software Mutationen im Virusgenom analysieren. Das hilft Wissenschaftlern dabei, die Mutationen zu identifizieren, die für die Auslösung der Erkrankung und die Übertragung der Viren verantwortlich sind. Die Plattform soll Forschungsgruppen motivieren, ihre Daten schnell zu teilen.
„Ich denke, dass bei der Bekämpfung von Krankheitsausbrüchen Open Science und Datenaustausch viel bewirken kann. Wir wollen eine Plattform bieten, die Einblick in Daten gewährt, ohne dem Urheber der Daten die Anerkennung der Erstveröffentlichung zu nehmen“, sagt Bedford.
Der Open Science Prize wird von einem Zusammenschluss aus dem US Wellcome Trust, den Nationalen Gesundheitsinstituten der USA (NIH) und dem Howard Hughes Medical Institute verliehen, um den offenen Austausch von Daten zu fördern und so die biomedizinische Forschung und deren Anwendungen für die Gesundheit voranzubringen.
Es gibt zwei Runden der Preisvergabe. In der ersten, nun abgeschlossenen Phase haben sich internationale Teams um die Preisgelder beworben. Gefördert wird die Umsetzung von Ideen für neue Produkte oder Anwendungen bis zum Prototypen. Bis zu sechs Projekte werden mit 80 000 US-Dollar finanziert. Die zweite Phase gewinnt das Projekt aus Phase eins, das den vielversprechendsten Prototypen entwickelt. Die zweite Phase ist mit 230 000 US-Dollar dotiert.